Kapitel 4 R, zweiter Blick
Benötigte R-Pakete für dieses Kapitel:
4.1 Lernsteuerung
4.2 Objekttypen in R
Näheres zu Objekttypen findet sich in Sauer (2019), Kap. 5.2.
4.2.1 Überblick
In R ist praktisch alles ein Objekt. Ein Objekt meint ein im Computerspeicher repräsentiertes Ding, etwa eine Tabelle.
Vektoren und Dataframes (Tibbles) sind die vielleicht gängigsten Objektarten in R (vgl. Abb. 4.1, aus Sauer (2019)).
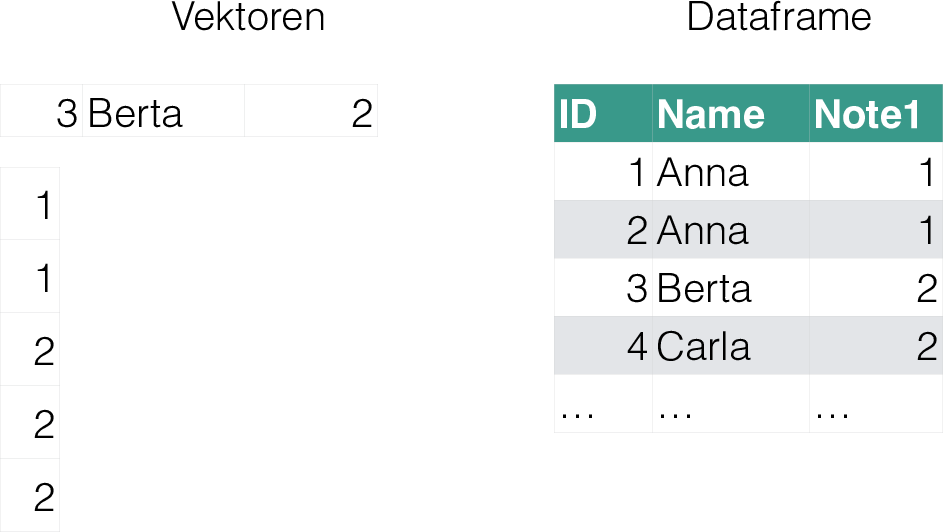
Abbildung 4.1: Zentrale Objektarten in R
Es gibt in R keine (Objekte für) Skalare (einzelne Zahlen). Stattdessen nutzt R Vektoren der Länge 1.
Ein nützliches Schema stammt aus Wickham and Grolemund (2016), s. Abb. 4.2.
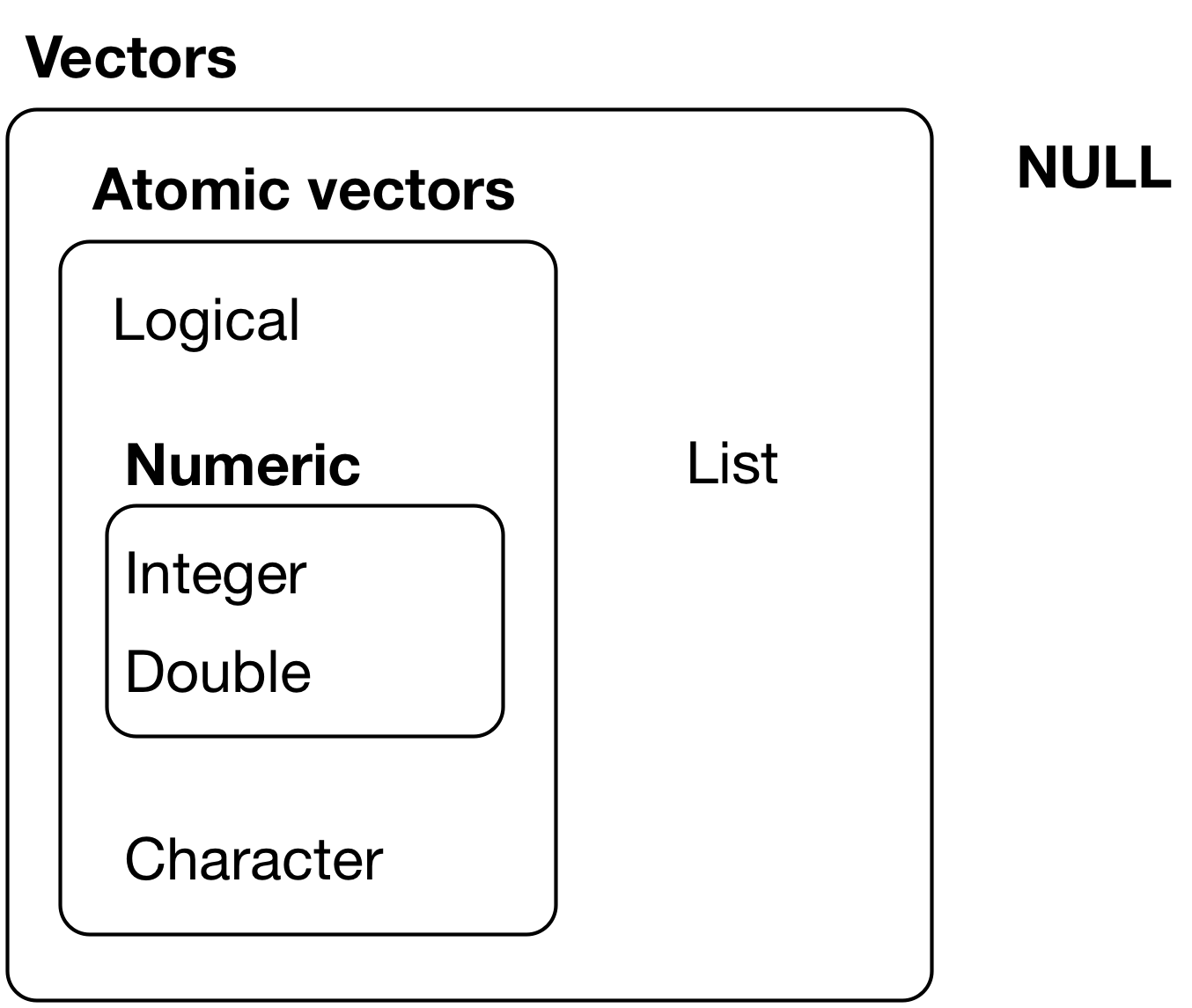
Abbildung 4.2: Objektarten hierarchisch gegliedert
4.2.2 Taxonomie
Unter homogenen Objektiven verstehen wir Datenstrukturen, die nur eine Art von Daten (wie Text oder Ganze Zahlen) fassen. Sonstige Objekte nennen wir heterogen.
- Homogene Objekte
- Vektoren
- Matrizen
- Heterogen
- Liste
- Dataframes (Tibbles)
4.2.2.1 Vektoren
Vektoren sind insofern zentral in R, als dass die übrigen Datenstrukturen auf ihnen aufbauen, vgl. Abb. 4.3 aus Sauer (2019).
Reine (atomare) Vektoren in R sind eine geordnete Liste von Daten eines Typs.
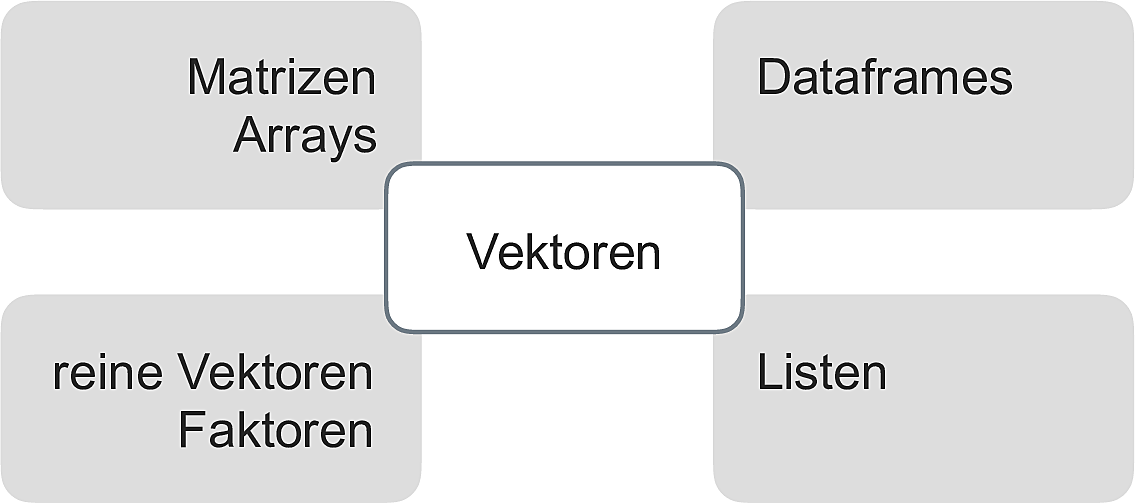
Abbildung 4.3: Vektoren stehen im Zentrum der Datenstrukturen in R
ein_vektor <- c(1, 2, 3)
noch_ein_vektor <- c("A", "B", "C")
logischer_vektor <- c(TRUE, FALSE, TRUE)Mit str() kann man sich die Struktur eines Objektsausgeben lassen:
str(ein_vektor)## num [1:3] 1 2 3
str(noch_ein_vektor)## chr [1:3] "A" "B" "C"
str(logischer_vektor)## logi [1:3] TRUE FALSE TRUEVektoren können von folgenden Typen sein:
- Kommazahlen (
double) genannt - Ganzzahlig (
integer, auch mitLfür Long abgekürzt) - Text (´character`, String)
- logische Ausdrücke (
logicaloderlgl) mitTRUEoderFALSE
Kommazahlen und Ganze Zahlen zusammen bilden den Typ numeric (numerisch) in R.
knitr::opts_chunk$set(echo = TRUE)Den Typ eines Vektors kann man mit typeof() ausgeben lassen:
typeof(ein_vektor)## [1] "double"4.2.2.2 Faktoren
Interessant:
str(sex)## Factor w/ 2 levels "Frau","Mann": 2 1 1Vertiefende Informationen findet sich in Wickham and Grolemund (2016).
4.2.2.3 Listen
eine_liste <- list(titel = "Einführung",
woche = 1,
datum = c("2022-03-14", "2202-03-21"),
lernziele = c("dies", "jenes", "und noch mehr"),
lehre = c(TRUE, TRUE, TRUE)
)
str(eine_liste)## List of 5
## $ titel : chr "Einführung"
## $ woche : num 1
## $ datum : chr [1:2] "2022-03-14" "2202-03-21"
## $ lernziele: chr [1:3] "dies" "jenes" "und noch mehr"
## $ lehre : logi [1:3] TRUE TRUE TRUE4.2.2.4 Tibbles
Für tibble() brauchen wir tidyverse:
studentis <-
tibble(
name = c("Anna", "Berta"),
motivation = c(10, 20),
noten = c(1.3, 1.7)
)
str(studentis)## tibble [2 × 3] (S3: tbl_df/tbl/data.frame)
## $ name : chr [1:2] "Anna" "Berta"
## $ motivation: num [1:2] 10 20
## $ noten : num [1:2] 1.3 1.74.2.3 Indizieren
Einen Teil eines Objekts auszulesen, bezeichnen wir als Indizieren.
4.2.3.1 Reine Vektoren
Zur Erinnerung:
str(ein_vektor)## num [1:3] 1 2 3
ein_vektor[1]## [1] 1
ein_vektor[c(1,2)]## [1] 1 2Aber nicht so:
ein_vektor[1,2]## Error in ein_vektor[1, 2]: incorrect number of dimensionsMan darf Vektoren auch wie Listen ansprechen, also eine doppelte Eckklammer zum Indizieren verwenden
ein_vektor[[2]]## [1] 2Der Grund ist, dass Listen auch Vektoren sind, nur eben ein besonderer Fall eines Vektors:
is.vector(eine_liste)## [1] TRUEWas passiert, wenn man bei einem Vektor der Länge 3 das 4. Element indiziert?
ein_vektor[4]## [1] NAEin schnödes NA ist die Antwort. Das ist interessant:
Wir bekommen keine Fehlermeldung, sondern den Hinweis,
das angesprochene Element sei leer bzw. nicht verfügbar.
In Sauer (2019), Kap. 5.3.1 findet man weitere Indizierungsmöglichkeiten für reine Vektoren.
4.2.3.2 Listen
## List of 5
## $ titel : chr "Einführung"
## $ woche : num 1
## $ datum : chr [1:2] "2022-03-14" "2202-03-21"
## $ lernziele: chr [1:3] "dies" "jenes" "und noch mehr"
## $ lehre : logi [1:3] TRUE TRUE TRUEListen können wie Vektoren, also mit [ ausgelesen werden.
Dann wird eine Liste zurückgegeben.
eine_liste[1]## $titel
## [1] "Einführung"
eine_liste[2]## $woche
## [1] 1Das hat den technischen Hintergrund, dass Listen als eine bestimmte Art von Vektoren implementiert sind.
Mann kann auch die “doppelte Eckklammer”, [[ zum Auslesen verwenden;
dann wird anstelle einer Liste die einfachere Struktur eines Vektors zurückgegeben:
eine_liste[[1]]## [1] "Einführung"Man könnte sagen, die “äußere Schicht” des Objekts, die Liste, wird abgeschält, und man bekommnt die “innere” Schicht, den Vektor.
Mann die Elemente der Liste entweder mit ihrer Positionsnummer (1, 2, …) oder, sofern vorhanden, ihren Namen ansprechen:
eine_liste[["titel"]]## [1] "Einführung"Dann gibt es noch den Dollar-Operator, mit dem Mann benannte Elemente von Listen ansprechen kann:
eine_liste$titel## [1] "Einführung"Man kann auch tiefer in eine Liste hinein indizieren.
Sagen wir, uns interessiert das 4. Element der Liste eine_liste -
und davon das erste Element.
Das geht dann so:
eine_liste[[4]][[1]] ## [1] "dies"Eine einfachere Art des Indizierens von Listen bietet die Funktion pluck(), aus dem Paket purrr,
das Hilfen für den Umgang mit Listen bietet.
pluck(eine_liste, 4)## [1] "dies" "jenes" "und noch mehr"Und jetzt aus dem 4. Element das 1. Element:
pluck(eine_liste, 4, 1)## [1] "dies"Probieren Sie mal, aus einer Liste der Länge 5 das 6. Element auszulesen:
## [1] 5
eine_liste[[6]]## Error in eine_liste[[6]]: subscript out of boundsUnser Versuch wird mit einer Fehlermeldung quittiert.
Sprechen wir die Liste wie einen (atomaren) Vektor an,
bekommen wir hingegen ein NA bzw. ein NULL:
eine_liste[6]## $<NA>
## NULL4.2.3.3 Tibbles
Tibbles lassen sich sowohl wie ein Vektor als auch wie eine Liste indizieren.
studentis[1]## # A tibble: 2 × 1
## name
## <chr>
## 1 Anna
## 2 BertaDie Indizierung eines Tibbles mit der einfachen Eckklammer liefert einen Tibble zurück.
studentis["name"]## # A tibble: 2 × 1
## name
## <chr>
## 1 Anna
## 2 BertaMit doppelter Eckklammer bekommt man, analog zur Liste, einen Vektor zurück:
studentis[["name"]]## [1] "Anna" "Berta"Beim Dollar-Operator kommt auch eine Liste zurück:
studentis$name## [1] "Anna" "Berta"4.2.4 Weiterführende Hinweise
- Tutorial zum Themen Indizieren von Listen von Jenny BC.
4.3 Datensätze von lang nach breit umformatieren
Manchmal findet man Datensätze im sog. langen Format vor, manchmal im breiten.
In der Regel müssen die Daten “tidy” sein, was meist dem langen Format entspricht, vgl. Abb. 4.4 aus Sauer (2019).
knitr::include_graphics("img/gather_spread.png")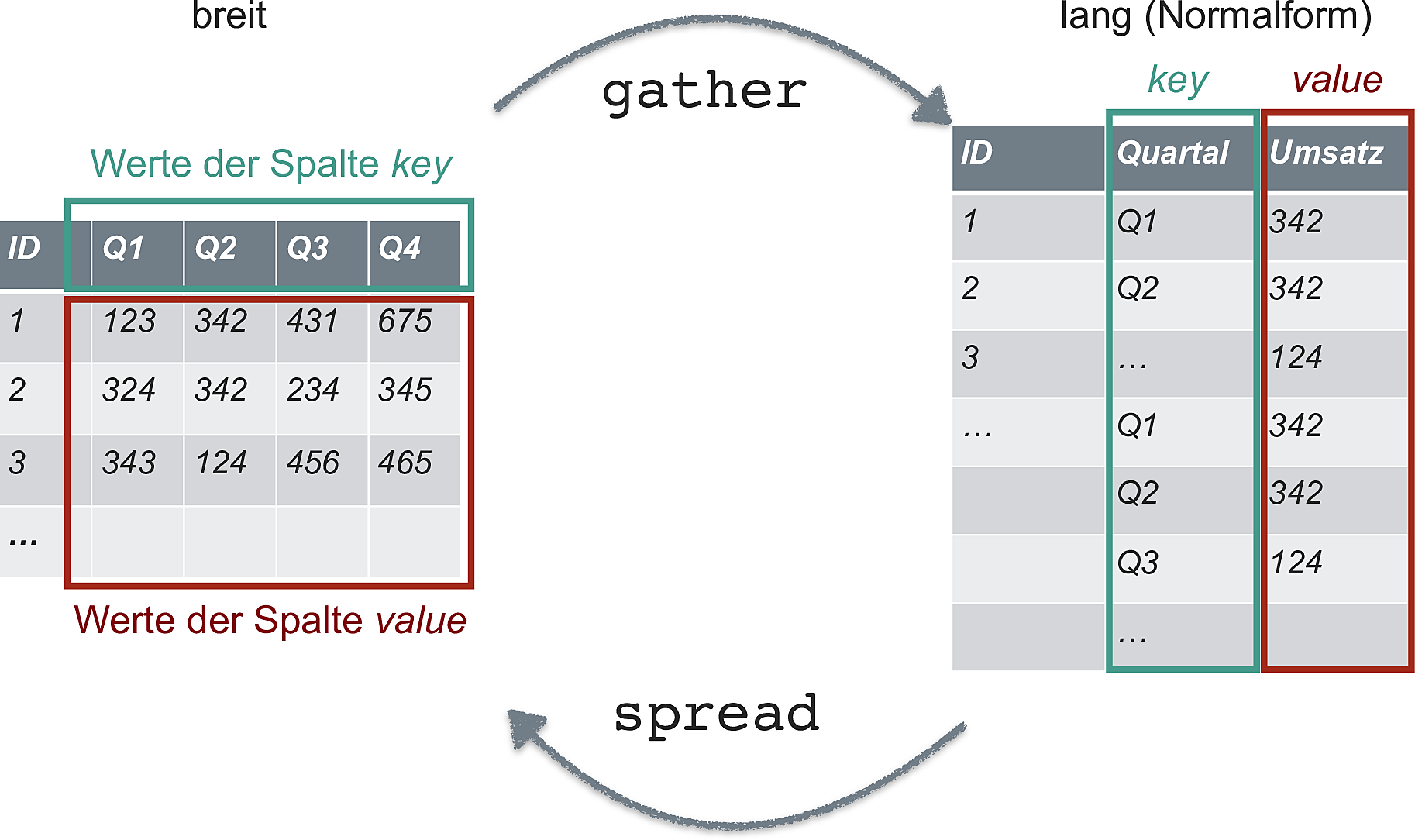
Abbildung 4.4: Von lang nach breit und zurück
In einer neueren Version des Tidyverse werden diese beiden Befehle umbenannt bzw. erweitert:
Weitere Informationen findet sich in Wickham and Grolemund (2016), in diesem Abschnitt, 12.3.
4.4 Funktionen
Eine Funktion kann man sich als analog zu einer Variable vorstellen. Es ist ein Objekt, das nicht Daten, sondern Syntax beinhaltet, vgl. Abb. 4.5 aus Sauer (2019).
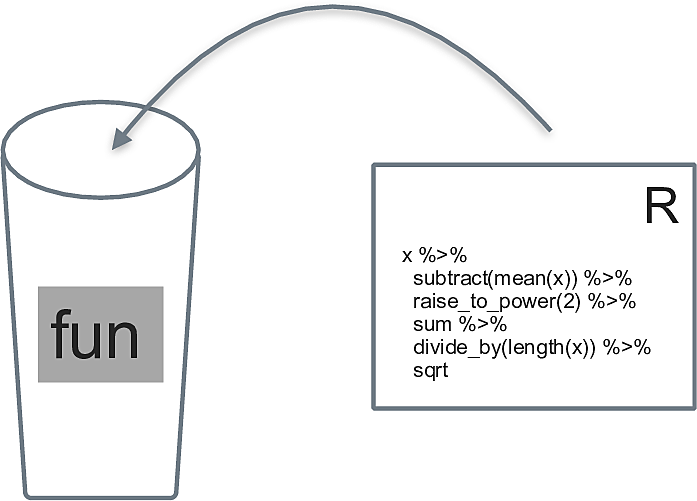
Abbildung 4.5: Sinnbild einer Funktion
mittelwert(c(1, 2, 3))## [1] 2Weitere Informationen finden sich in Kapitel 19 in Wickham and Grolemund (2016). Alternativ findet sich ein Abschnitt dazu (28.1) in Sauer (2019).
4.5 Wiederholungen programmieren
Häufig möchte man eine Operation mehrfach ausführen. Ein Beispiel wäre die Anzahl der fehlenden Werte pro Spalte auslesen. Natürlich kann man die Abfrage einfach häufig tippen, nervt aber irgendwann. Daher braucht’s Strukturen, die Wiederholungen beschreiben.
Dafür gibt es verschiedene Ansätze.
4.5.1 across()
Handelt es sich um Spalten von Tibbles, dann bietet sich die Funktion across(.col, .fns) an.
across wendet eine oder mehrere Funktionen (mit .fns bezeichnet) auf die Spalten .col an.
Das erklärt sich am besten mit einem Beispiel:
Natürlich hätte man in diesem Fall auch anders vorgehen können:
mtcars %>%
summarise(across(.cols = everything(),
.fns = mean))## mpg cyl disp hp drat wt qsec vs am
## 1 20.09062 6.1875 230.7219 146.6875 3.596563 3.21725 17.84875 0.4375 0.40625
## gear carb
## 1 3.6875 2.8125Möchte man der Funktion .fns Parameter übergeben, so nutzt man diese Syntax (“Purrr-Lambda”):
mtcars %>%
summarise(across(.cols = everything(),
.fns = ~ mean(., na.rm = TRUE)))## mpg cyl disp hp drat wt qsec vs am
## 1 20.09062 6.1875 230.7219 146.6875 3.596563 3.21725 17.84875 0.4375 0.40625
## gear carb
## 1 3.6875 2.8125
4.5.2 map()
map() ist eine Funktion aus dem R-Paket purrr und Teil des Tidyverse.
map(x, f) wenden die Funktion f auf jedes Element von x an.
Ist x ein Tibble, so wird f demnach auf jede Spalte von x angewendet (“zugeordnet”, daher map), vgl. Abb. 4.6 aus Sauer (2019).
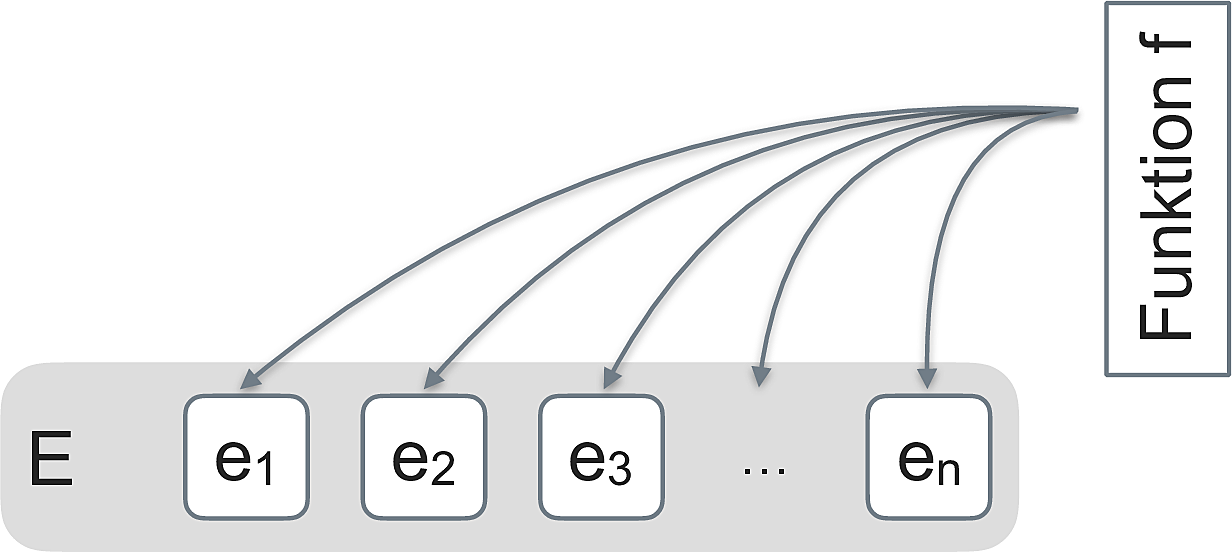
Abbildung 4.6: Sinnbild für map
Hier ein Beispiel-Code:
## $mpg
## [1] 20.09062
##
## $cyl
## [1] 6.1875
##
## $disp
## [1] 230.7219Möchte man der gemappten Funktion Parameter übergeben, nutzt man wieder die “Kringel-Schreibweise”:
## $mpg
## [1] 20.09062
##
## $cyl
## [1] 6.1875
##
## $disp
## [1] 230.72194.6 Listenspalten
4.6.1 Wozu Listenspalten?
Listenspalten sind immer dann sinnvoll, wenn eine einfache Tabelle nicht komplex genug für unsere Daten ist.
Zwei Fälle stechen dabei ins Auge:
- Unsere Datenstruktur ist nicht rechteckig
- In einer Zelle der Tabelle soll mehr als ein einzelner Wert stehen: vielleicht ein Vektor, eine Liste oder eine Tabelle
Der erstere Fall (nicht reckeckig) ließe sich noch einfach lösen,
in dem man mit NA auffüllt.
Der zweite Fall verlangt schlichtweg nach komplexeren Datenstrukturen.
Kap. 25.3 aus Wickham and Grolemund (2016) bietet einen guten Einstieg in das Konzept von Listenspalten (list-columns) in R.
4.6.2 Beispiele für Listenspalten
4.6.2.1 tidymodel
Wenn wir mit tidymodels arbeiten,
werden wir mit Listenspalten zu tun haben.
Daher ist es praktisch, sich schon mal damit zu beschäftigen.
Hier ein Beispiel für eine \(v=3\)-fache Kreuzvalidierung:
library(tidymodels)
mtcars_cv <-
vfold_cv(mtcars, v = 3)
mtcars_cv## # 3-fold cross-validation
## # A tibble: 3 × 2
## splits id
## <list> <chr>
## 1 <split [21/11]> Fold1
## 2 <split [21/11]> Fold2
## 3 <split [22/10]> Fold3Betrachten wir das Objekt mtcars_cv näher.
Die Musik spielt in der 1. Spalte.
Lesen wir den Inhalt der 1. Spalte, 1 Zeile aus (nennen wir das mal “Position 1,1”):
pos11 <- mtcars_cv[[1]][[1]]
pos11## <Analysis/Assess/Total>
## <21/11/32>In dieser Zelle findet sich eine Aufteilung des Komplettdatensatzes in den Analyseteil (Analysis sample) und den Assessmentteil (Assessment Sample).
Schauen wir jetzt in dieses Objekt näher an.
Das können wir mit str() tun.
str() zeigt uns die Strktur eines Objekts.
str(pos11)## List of 4
## $ data :'data.frame': 32 obs. of 3 variables:
## ..$ mpg : num [1:32] 21 21 22.8 21.4 18.7 18.1 14.3 24.4 22.8 19.2 ...
## ..$ cyl : num [1:32] 6 6 4 6 8 6 8 4 4 6 ...
## ..$ disp: num [1:32] 160 160 108 258 360 ...
## $ in_id : int [1:21] 2 6 8 9 10 11 12 14 15 16 ...
## $ out_id: logi NA
## $ id : tibble [1 × 1] (S3: tbl_df/tbl/data.frame)
## ..$ id: chr "Fold1"
## - attr(*, "class")= chr [1:2] "vfold_split" "rsplit"Oh! pos11 ist eine Liste, und zwar eine durchaus komplexe.
Wir müssen erkennen,
dass in einer einzelnen Zelle dieses Dataframes viel mehr steht,
als ein Skalar bzw. ein einzelnes, atomares Element.
Damit handelt es sich bei Spalte 1 dieses Dataframes (mtcars_cv) also um eine Listenspalte.
Üben wir uns noch etwas im Indizieren.
Sprechen wir in pos11 das erste Element an (data) und davon das erste Element:
pos11[["data"]][[1]]## [1] 21.0 21.0 22.8 21.4 18.7 18.1 14.3 24.4 22.8 19.2 17.8 16.4 17.3 15.2 10.4
## [16] 10.4 14.7 32.4 30.4 33.9 21.5 15.5 15.2 13.3 19.2 27.3 26.0 30.4 15.8 19.7
## [31] 15.0 21.4Wir haben hier die doppelten Eckklammern benutzt, um den “eigentlichen” oder “inneren” Vektor zu bekommen, nicht die “außen” herumgewickelte Liste. Zur Erinnerung: Ein Dataframe ist ein Spezialfall einer Liste, also auch eine Liste, nur eine mit bestimmten Eigenschaften.
Zum Vergleich indizieren wir mal mit einer einfachen Eckklammer:
## mpg
## Mazda RX4 21.0
## Mazda RX4 Wag 21.0
## Datsun 710 22.8
## Hornet 4 Drive 21.4
## Hornet Sportabout 18.7
## Valiant 18.1Mit pluck() bekommen wir das gleiche Ergebnis,
nur etwas komfortabler,
da wir keine Eckklammern tippen müssen:
pluck(pos11, "data", 1, 1)## [1] 21Wie man sieht, können wir beliebig tief in das Objekt hineinindizieren.
4.6.2.2 Kurs DataScience1
Ein Kurs, wie dieser, kann anhand einer “Deskriptoren” wie Titel der Inhalte, Lernziele, Literatur und so weiter zusammmengefasst werden. Diese Deskriptoren kann man wiederum jeder Kurswoche oder jedem Kursabschnitt zuordnen, so dass eine zweidimensionale Struktur resultiert. Eine Tabelle, einfach gesagt, etwa so:
tibble::tribble(
~Nr, ~Titel, ~Literatur, ~Aufgaben,
1L, "Titel1", "Literatur1", "Aufgaben1",
2L, "Titel2", "Literatur2", "Aufgaben2",
3L, "Titel3", "Literatur3", "Aufgaben3"
) %>%
gt::gt()| Nr | Titel | Literatur | Aufgaben |
|---|---|---|---|
| 1 | Titel1 | Literatur1 | Aufgaben1 |
| 2 | Titel2 | Literatur2 | Aufgaben2 |
| 3 | Titel3 | Literatur3 | Aufgaben3 |
Wie man sieht, entspricht jede Spalte einem Deskriptor des Kurses, und jede Zeile entspricht einem Thema (oder Woche oder Abschnitt) des Kurses.
Jetzt ist es nur so, dass einzelne Zellen dieser Tabelle nicht aus nur einem Element bestehen. So könnte etwa “Aufgaben1” aus mehreren Aufgaben bestehen, die jeweils wiederum aus mehreren (Text-)Elementen bestehen. Oder “Literatur2” besteht vielleicht aus zwei Literaturquellen.
Kurz gesagt, wir brauchen eine Tabelle, die erlaubt, in einer Zelle mehr als ein einzelnes Element zu packen. Listenspalten erlauben das.
Schauen wir uns die “Mastertabelle” dieses Kurses an zur Illustration.
Zunächst sourcen wir die nötigen Funktionen.
source("https://raw.githubusercontent.com/sebastiansauer/Lehre/main/R-Code/render-course-sections.R")In Ihrem Environment sollten Sie jetzt die gesourcten Funktionen sehen.
Mit Klick auf den Funktionsnamen können Sie diese Funktionen auch betrachten.
Die Deskriptoren des Kurses speisen sich aus zwei Textdateien, gespeichert im sog. YAML-Format, ein einfaches Textformat, und hier nicht weiter von Belang.
Zum einen eine Datei mit den Datumsangaben:
course_dates_file <- "https://raw.githubusercontent.com/sebastiansauer/datascience1/main/course-dates.yaml"Zum anderen eine Datei mit den Deskriptoren, die unabhängig vom Datum sind:
content_file <- "https://raw.githubusercontent.com/sebastiansauer/datascience1/main/_modul-ueberblick.yaml"Im Githup-Repo dieses Kurses können Sie die Dateien komfortabel betrachten.
Die “Mastertabelle” kann man mit folgender Funktion erstellen:
mastertable <- build_master_course_table(
course_dates_file = course_dates_file,
content_file = content_file)Betrachten Sie die Tabelle in Ruhe! Sie werden sehen, dass einige Spalten komplex sind, also mehr als nur einen einzelnen Wert enthalten:
mastertable[["Vorbereitung"]][[1]]## [1] "Lesen Sie die Hinweise zum Modul."
## [2] "Installieren (oder Updaten) Sie die für dieses Modul angegeben Software."
## [3] "Lesen Sie die Literatur."Gerade haben wir aus dem Objekt mastertable, ein Dataframe,
die Spalte mit dem Namen Vorbereitung ausgelesen und aus dieser Spalte das erste erste Element.
Dieses erste Element ist ein Textvektor der Länge 3.
Daraus könnten wir z.B. das zweite Element auslesen:
mastertable[["Vorbereitung"]][[1]][2]## [1] "Installieren (oder Updaten) Sie die für dieses Modul angegeben Software."Was würde passieren, wenn wir anstelle der doppelten Eckklammer einfache Eckklammern verwenden würden?
## [1] "tbl_df" "tbl" "data.frame"## [1] "list"Das macht noch keinen großen Unterschied, aber schauen wir mal weiter.
Wenn wir das erste Element der Spalte “Vorbereitung” mit doppelter Eckklammer ansprechen, bekommen wir einen Text-Vektor (der Länge drei) zurück.
mastertable[["Vorbereitung"]][[1]]## [1] "Lesen Sie die Hinweise zum Modul."
## [2] "Installieren (oder Updaten) Sie die für dieses Modul angegeben Software."
## [3] "Lesen Sie die Literatur."Jetzt können wir, wie oben getan, diese einzelnen Elemente ansprechen.
Aber: Wenn wir das erste Element der Spalte “Vorbereitung” mit einfacher Eckklammer ansprechen, bekommen wir eine Liste mit einem Element zurück.
mastertable[["Vorbereitung"]][1]## [[1]]
## [1] "Lesen Sie die Hinweise zum Modul."
## [2] "Installieren (oder Updaten) Sie die für dieses Modul angegeben Software."
## [3] "Lesen Sie die Literatur."Wir können also nicht (ohne weiteres “Abschälen”) z.B. das zweite Element des Text-Vektors (“Installieren Sie…”) auslesen:
## NULLWenn Sie sich über pluck() wundern,
Sie hätten synonym auch schreiben können:
mastertable[["Vorbereitung"]][1][2]## [[1]]
## NULLDa die Liste nur aus einem Element besteht, könnten wir z.B. nicht das zweite Element der Liste ansprechen:
mastertable[["Vorbereitung"]][1][[2]]## Error in mastertable[["Vorbereitung"]][1][[2]]: subscript out of boundsHaben wir aber die doppelte Eckklammer verwendet, so bekommen wir einen Vektor der Länge drei zurück (vom Typ Text), und daher können wir die Elemente 1 bis 3 ansprechen:
mastertable[["Vorbereitung"]][[1]][2]## [1] "Installieren (oder Updaten) Sie die für dieses Modul angegeben Software."Dabei ist es egal, ob Sie einfache oder doppelte Eckklammern benutzen, da Listen auch Vektoren sind.
4.6.3 Programmieren mit dem Tidyverse
Das Programmieren mit dem Tidyvers ist nicht ganz einfach und hier nicht näher ausgeführt. Eine Einführung findet sich z.B.
- Tidyeval in fünf Minuten (Video)
- In Kapiteln 17-21 in Advanced R, 2nd Ed
- Ein Überblicksdiagramm findet sich hier Quelle.
4.7 R ist schwierig
Manche behaupten, R sei ein Inferno.
Zum Glück gibt es auch aufmunternde Stimmen:
praise::praise()## [1] "You are smashing!"Hat jemand einen guten Rat für uns? Vielleicht ist der häufigste Rate, dass man die Dokumentation lesen solle.
4.8 Aufgaben
-
Aufgabe
Schreiben Sie eine Funktion, mit folgendem Algorithmus, wobei ein beliebiger Vektor als Eingabe verlangt wird.
- Zähle die Anzahl verschiedener Werte.
- Wenn es nur zwei verschiedene Werte gibt, gebe
TRUEzurück, ansonstenFALSE.
Hinweise:
- Wählen Sie einen treffenden Namen für Ihre Funktion (nutzen Sie am besten ein konsistentes Namensschema).
- Wichtigster Tipp: Googeln :-)
- Verschiedene Werte eines Vektors gibt die Funktion
unique()zurück. - Vermutlich gibt es schon viele Lösungen (Implementierungen) für diese Funktion. Ist nur als Übung gedacht :-)
Lösunghas_two_levels <- function(vec){ # input: vector of any type # value: number of unique values (double) tmp <- length(unique(vec)) tmp == 2 }levelsist ein Ausdruck, der nahelegt, dass es sich um verschiedene Werte (“Ausprägungen” oder “Stufen”) handelt.Alternativ könnte man die Funktion auch so schreiben:
has_two_values <- function(vec){ # input: vector of any type # value: number of unique values (double) step1 <- unique(vec) step2 <- length(step1) step3 <- if(step2 == 2) { out <- TRUE } else { out <- FALSE } out <- step2 return(out) }Das ist expliziter, aber länger.
Wenn man es genau nimmt, heißt binär, dass es nur die Werte
0und1gibt. Das ist ein strengeres Kriterium, wie dass es zwei beliebigen verschiedene Werte gibt (s. unten dazu ein Vorschlag).Testen wir unsere Funktion, Test 1:
x <- c(1,2, 3, 3, 3, 1) x2 <- c("A", "B") has_two_levels(x2)## [1] TRUEhas_two_levels(x)## [1] FALSETest 2:
data(mtcars)Wir wenden unsere neue Funktion mit Tidyverse-Methoden an:
mtcars %>% summarise(am_has_two_values = has_two_levels(am), mpg_has_two_values = has_two_levels(mpg))## am_has_two_values mpg_has_two_values ## 1 TRUE FALSEBonus!
Verwenden Sie
across()(dplyr), um alle Spalten vonmtcarsmithas_two_levels()zu überprüfen.mtcars %>% summarise(across(everything(), has_two_levels))## mpg cyl disp hp drat wt ## 1 FALSE FALSE FALSE FALSE FALSE FALSE ## qsec vs am gear carb ## 1 FALSE TRUE TRUE FALSE FALSEKann man auch so schreiben:
mtcars %>% summarise(across(everything(), ~ has_two_levels(.)))## mpg cyl disp hp drat wt ## 1 FALSE FALSE FALSE FALSE FALSE FALSE ## qsec vs am gear carb ## 1 FALSE TRUE TRUE FALSE FALSEBonus-Bonus:
So könnte man eine Funktion schreiben, die prüft, ob die Ausprägungen eines Vektors binär sind, d.h. nur
0oder1:is_binary <- function(var){ return(all(var %in% c(0,1))) } -
Aufgabe
Schreiben Sie eine Funktion zur Berechnung der Anzahl der fehlenden Werte in einem (numerischen) Vektor!
Hinweise:
- Wählen Sie einen treffenden Namen für Ihre Funktion (nutzen Sie am besten ein konsistentes Namensschema).
Lösungna_n <- function(num_vec) { # input: num. vector (int or double) # value: count of missing values (double) if (!is.numeric(num_vec)) stop("Numeric input is needed!") out <- sum(is.na(num_vec)) return(out) }Test:
x <- c(1,2, NA, NA) x2 <- c("A", "B", NA) na_n(x)## [1] 2Bei
x2sollte ein Fehler aufgeworfen werden:na_n(x2)## Error in na_n(x2): Numeric input is needed!Gut!
Testen wir weiter, jetzt mit dem Datensatz
mtcars:mtcars[1, c(1,2,3,4)] <- NA # Zeilen/Spalte mtcars %>% summarise(mpg_na_n = na_n(mpg))## mpg_na_n ## 1 1BONUS!
Verwenden Sie
across(), um die fehlenden Werte in allen Spalten vonmtcarszu zählen.Wer schnell ist, der nehme gerne
nycflights13::flights(aus dem Paket nycflights13 oder per CSV-Datei aus geeigneter Stelel aus dem Internet. Meistens geht es schneller, die Daten aus einem Paket zu laden mitdata(flights)nachdem manlibrary(nycflights13)geschrieben hat).mtcars %>% summarise(across(everything(), na_n))## mpg cyl disp hp drat wt qsec vs am ## 1 1 1 1 1 0 0 0 0 0 ## gear carb ## 1 0 0Mit
pivot_longer()ist es häufig übersichtlicher bzw. besser für weitere Bearbeitungsschritte, wie das folgende Beispiel zeigt:mtcars %>% summarise(across(everything(), na_n)) %>% pivot_longer(everything()) %>% filter(value > 0)## # A tibble: 4 × 2 ## name value ## <chr> <int> ## 1 mpg 1 ## 2 cyl 1 ## 3 disp 1 ## 4 hp 1flights:data(flights, package = "nycflights13") flights %>% select(where(is.numeric)) %>% summarise(across(everything(), na_n)) %>% pivot_longer(everything()) %>% arrange(-value) %>% # Sortiere absteigend nach Anzahl der fehlenden Werte filter(value > 0) # Zeige nur Variablen mit fehlenden Werten## # A tibble: 5 × 2 ## name value ## <chr> <int> ## 1 arr_delay 9430 ## 2 air_time 9430 ## 3 arr_time 8713 ## 4 dep_time 8255 ## 5 dep_delay 8255 -
Aufgabe
Erstellen Sie für jede Variable aus
mtcarsein Histogramm!Hinweise:
- Nutzen Sie eine Wiederholungsstruktur. Schreiben Sie prägnante Syntax.
- Optional: Lassen Sie dichotome (zweiwertige) Variablen aus.
- Hier (und an vielen anderen Stellen im Netz) finden Sie Tipps.
Lösunghas_two_levels <- function(vec){ # input: vector of any type # value: number of unique values (double) tmp <- length(unique(vec)) tmp == 2 }mtcars_hist <- function(col){ mtcars %>% ggplot(aes(x = col)) + geom_histogram() }mtcars %>% select(where(is.numeric)) %>% select(where(negate(has_two_levels))) %>% # `!` zum Negieren geht leider nicht map(mtcars_hist)## $mpg ## ## $cyl ## ## $disp ## ## $hp ## ## $drat ## ## $wt ## ## $qsec ## ## $gear ## ## $carb