graph LR ind1 --> lonely ind2 --> lonely u --> lonely
einsamkeit-modellierung
1 Fallstudie zur Einsamkeitsinduktion
In dieser Fallstudie wird eine studentische Studie vorgestellt, die untersucht, inwieweit Einsamkeit induziert werden kann und welche Effekte die Einsamkeitsinduktion auf das aktuelle Befinden hat.
Das Design ist reichhaltig und lässt viele Analysen zu. Bei diesem Post liegt der Fokus auf der Modellierung der Forschungsfrage.
1.1 Forschungsfrage und Hypothesen
Hat die Induktion (ind) von Einsamkeit einen Effekt auf die wahrgenommenen Einsamkeit (lonely) ?
Insgesamt wird zwei Mal Einsamkeit induziert:
H1: Die Induktion 1 (ind1) verringert die Einsamkeit (Geschichte über Einsamkeit oder neutral)
H2: Die Induktion 2 (ind2) verringert die Einsamkeit (“einsame” Musik oder neutral)
1.2 DAG (Kausalgraph)
Es handelt sich um eine Kausalanalyse, da man am Effekt der Intervention auf die Einsamkeit interessiert ist.
1.3 Forschungsdesign
UV1: Einsamkeitsinduktion 1 (Geschichte)
UV2: Einsamkeitsinduktion 2 (Musik)
AV: Einsamkeit
2 Hinweise
Beachten Sie die üblichen Hinweise des Datenwerks.
3 Setup
4 Daten importieren
url <- "https://github.com/sebastiansauer/Datenwerk/raw/refs/heads/main/posts/einsamkeit-modellierung/einsamkeit.csv"
d <- read_csv(url)Check:
glimpse(d)Rows: 44
Columns: 21
$ Proband_ID <dbl> 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 1…
$ Gruppe <dbl> 3, 2, 1, 4, 2, 1, 4, 3, 1, 2, 4, 3, 3, 1…
$ Geschichte <chr> "neutrale", "einsame", "neutrale", "eins…
$ Musik <chr> "neutrale", "neutrale", "einsame", "eins…
$ SAM_Pleasure_f1 <dbl> 7, 7, 9, 7, 7, 7, 5, 9, 5, 9, 7, 7, 7, 9…
$ SAM_Pleasure_f2 <dbl> 7, 7, 7, 5, 5, 7, 5, 7, 7, 9, 7, 7, 7, 9…
$ SAM_Pleasure_f3 <dbl> 9, 5, 5, 9, 7, 9, 5, 9, 9, 9, 3, 9, 7, 9…
$ SAM_Arousal_f1 <dbl> 3, 1, 3, 7, 5, 3, 3, 5, 5, 7, 3, 5, 1, 3…
$ SAM_Arousal_f2 <dbl> 5, 1, 1, 5, 3, 3, 3, 3, 3, 3, 3, 3, 1, 5…
$ SAM_Arousal_f3 <dbl> 1, 1, 3, 7, 5, 3, 3, 1, 3, 1, 1, 5, 3, 5…
$ SAM_Dominance_f1 <dbl> 5, 5, 9, 7, 7, 7, 5, 7, 3, 7, 3, 5, 7, 5…
$ SAM_Dominance_f2 <dbl> 5, 5, 7, 5, 3, 7, 3, 7, 5, 7, 7, 5, 7, 7…
$ SAM_Dominance_f3 <dbl> 5, 3, 3, 7, 3, 7, 3, 9, 5, 7, 7, 5, 7, 9…
$ Loneliness_mean_f1 <dbl> 1.4, 1.2, 2.2, 1.6, 2.2, 1.4, 2.4, 1.8, …
$ Loneliness_mean_f2 <dbl> 1.2, 1.8, 2.2, 1.6, 2.0, 1.2, 2.4, 1.4, …
$ Loneliness_mean_f3 <dbl> 1.0, 1.8, 2.0, 1.2, 2.0, 1.2, 2.4, 1.4, …
$ Godspeed_Anthropo_mean_f3 <dbl> 2.8, 2.0, 1.0, 2.8, 2.0, 3.8, 1.2, 3.4, …
$ Godspeed_Animacy_mean_f3 <dbl> 3.833333, 2.000000, 2.166667, 3.500000, …
$ Godspeed_Likeability_mean_f3 <dbl> 5.0, 2.8, 2.2, 4.4, 3.0, 4.4, 3.0, 4.8, …
$ Godspeed_Intelligence_mean_f3 <dbl> 3.8, 2.6, 2.6, 3.4, 2.6, 4.4, 2.8, 3.4, …
$ Godspeed_Safety_mean_f3 <dbl> 3.666667, 5.000000, 2.000000, 2.333333, …d |>
describe_distribution() |>
kable(digits = 2)| Variable | Mean | SD | IQR | Min | Max | Skewness | Kurtosis | n | n_Missing |
|---|---|---|---|---|---|---|---|---|---|
| Proband_ID | 22.50 | 12.85 | 22.50 | 1.0 | 44.00 | 0.00 | -1.20 | 44 | 0 |
| Gruppe | 2.50 | 1.13 | 2.50 | 1.0 | 4.00 | 0.00 | -1.38 | 44 | 0 |
| SAM_Pleasure_f1 | 7.23 | 1.24 | 1.50 | 5.0 | 9.00 | -0.07 | -0.29 | 44 | 0 |
| SAM_Pleasure_f2 | 6.59 | 1.70 | 2.00 | 3.0 | 9.00 | -0.53 | -0.06 | 44 | 0 |
| SAM_Pleasure_f3 | 7.50 | 1.56 | 2.00 | 3.0 | 9.00 | -0.79 | 0.11 | 44 | 0 |
| SAM_Arousal_f1 | 3.64 | 1.92 | 2.00 | 1.0 | 9.00 | 0.47 | 0.13 | 44 | 0 |
| SAM_Arousal_f2 | 3.68 | 2.02 | 2.00 | 1.0 | 9.00 | 0.53 | -0.16 | 44 | 0 |
| SAM_Arousal_f3 | 3.59 | 1.91 | 2.00 | 1.0 | 9.00 | 0.54 | 0.25 | 44 | 0 |
| SAM_Dominance_f1 | 5.68 | 1.88 | 2.00 | 1.0 | 9.00 | -0.22 | 0.57 | 44 | 0 |
| SAM_Dominance_f2 | 5.27 | 2.00 | 2.00 | 1.0 | 9.00 | -0.29 | -0.10 | 44 | 0 |
| SAM_Dominance_f3 | 5.18 | 1.92 | 3.50 | 1.0 | 9.00 | -0.02 | -0.13 | 44 | 0 |
| Loneliness_mean_f1 | 1.92 | 0.34 | 0.60 | 1.2 | 2.60 | -0.05 | -0.72 | 44 | 0 |
| Loneliness_mean_f2 | 1.95 | 0.38 | 0.60 | 1.2 | 2.80 | 0.12 | -0.41 | 44 | 0 |
| Loneliness_mean_f3 | 1.92 | 0.39 | 0.60 | 1.0 | 2.80 | 0.00 | 0.18 | 44 | 0 |
| Godspeed_Anthropo_mean_f3 | 2.20 | 0.79 | 1.15 | 1.0 | 4.00 | 0.37 | -0.46 | 44 | 0 |
| Godspeed_Animacy_mean_f3 | 2.96 | 0.75 | 0.96 | 1.0 | 4.17 | -0.70 | 0.14 | 44 | 0 |
| Godspeed_Likeability_mean_f3 | 3.83 | 0.76 | 1.00 | 2.0 | 5.00 | -0.44 | -0.22 | 44 | 0 |
| Godspeed_Intelligence_mean_f3 | 3.31 | 0.74 | 1.20 | 2.0 | 5.00 | 0.42 | -0.30 | 44 | 0 |
| Godspeed_Safety_mean_f3 | 3.92 | 0.77 | 1.25 | 2.0 | 5.00 | -0.59 | 0.09 | 44 | 0 |
5 Ablauf
graph LR Start --> Messung_t1 --> Intervention1 --> Messung_t2 --> Intervention2 --> Messung_t3 --> Ende
Die Intervention ist in diesem Fall die Induktion von Einsamkeit.
Es handelt sich um Between-Desing mit Vorher-Nachher-Messung, auch als “Veränderungsmessung” oder Veränderungsdesign bezeichnet.
Die Versuchspersonen durchlaufen nicht die gleichen Bedingungen, sondern verschiedene: einige Probanden sind in der Experimentalgruppe, andere in der Kontrollgruppe.
Schauen wir genauer zur ersten Intervention:
graph LR Messung_t1 --> Intervention1 --> Messung_t2
Es gibt zwei Gruppen, die Experimentalgruppe bekommt die Einsamkeit, die Kontrollgruppe die neutrale Bedingung:
graph LR Messung_t1 --> G1[Einsamkeits-Induktion] Messung_t1 --> G2[Kontrollgruppe] G1 --> Messung_t2 G2 --> Messung_t2
6 Berechnung der Deltavariablen
Bei Veränderungsdesigns muss man die Veränderung von einem Messzeitpunkt zum anderen berechnen, pro Person. Dazwischen muss eine Intervention liegen. Dieser Veränderung nennen wir delta, sie misst den kausalen Effekt der Intervention. Je größer das (mittlere) Delta zwischen den Gruppen, desto größer der Effekt.
d_delta <-
d |>
mutate(loneliness_delta1 = Loneliness_mean_f2 - Loneliness_mean_f1,
loneliness_delta2 = Loneliness_mean_f3 - Loneliness_mean_f2,
loneliness_delta3 = Loneliness_mean_f3 - Loneliness_mean_f1 )Visualisierung der Delta-Variablen:
graph LR Start --> Messung_t1 --> Intervention1 --> Messung_t2 --> Intervention2 --> Messung_t3 Messung_t2 -- delta1 --> Messung_t1 Messung_t3 -- delta2 --> Messung_t2 Messung_t3 -- delta3 --> Messung_t1
Puh, das ist unübersichtlich. Vielleicht ist die Berechnung der Delta-Variablen so übersichtlicher:
graph LR Messung_t2 -- delta1 --> Messung_t1 Messung_t3 -- delta2 --> Messung_t2 Messung_t3 -- delta3 --> Messung_t1
7 Modellierung
7.1 Delta 1
7.1.1 Modell
mod_delta1 <- stan_glm(loneliness_delta1 ~ Geschichte, data = d_delta)Mit lm würde das genau so funktionieren. Nur ohne die Ergebnisse einfach probabilistisch interpretieren zu können.
Ergebnisse:
parameters(mod_delta1) |>
print_md()| Parameter | Median | 95% CI | pd | Rhat | ESS | Prior |
|---|---|---|---|---|---|---|
| (Intercept) | 0.07 | (-0.04, 0.18) | 90.62% | 1.000 | 3225 | Normal (0.02 +- 0.63) |
| Geschichteneutrale | -0.10 | (-0.26, 0.06) | 89.75% | 1.000 | 3568 | Normal (0.00 +- 1.25) |
Die Nullhypothese kann nicht verworfen werden.
7.1.2 Visualisierung
plot(parameters(mod_delta1))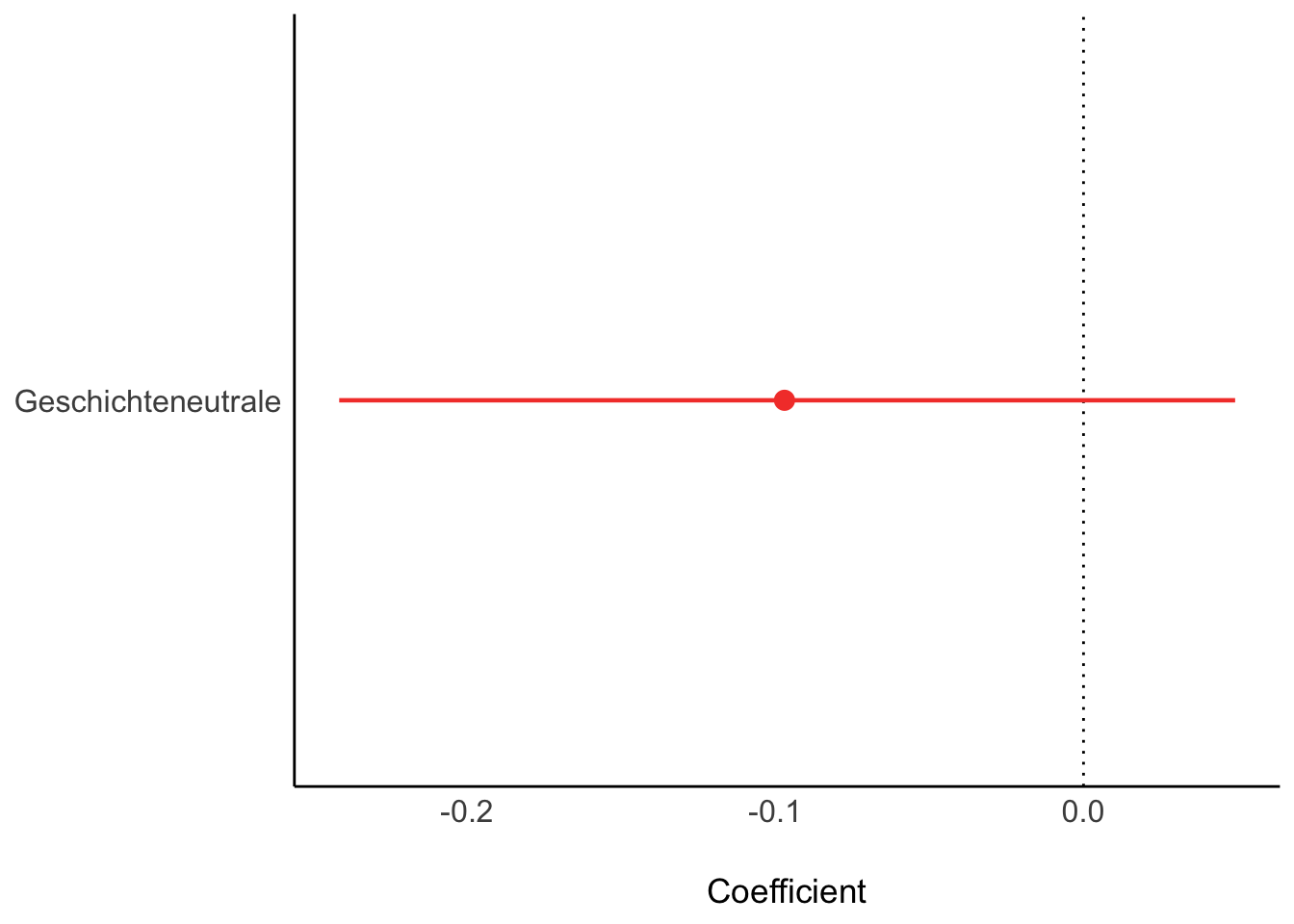
7.1.3 Modellgüte
r2(mod_delta1)# Bayesian R2 with Compatibility Interval
Conditional R2: 0.038 (95% CI [3.723e-08, 0.173])Tja, die Intervention war leider nicht so stark. Aber so ist es nun Mal im harten Forscherleben …
mod_delta1_rope <- rope(mod_delta1)
plot(mod_delta1_rope)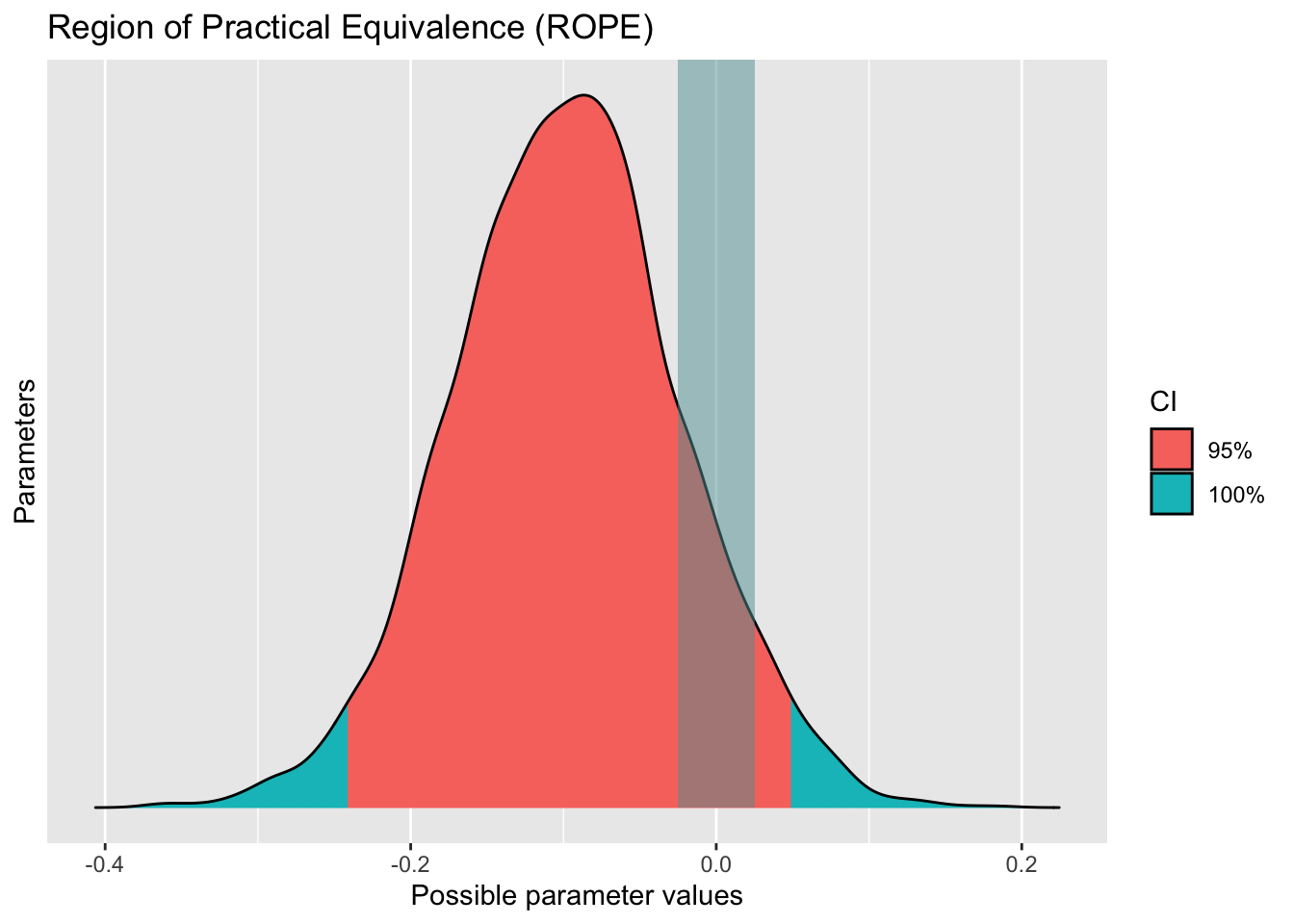
7.2 Delta 2 und 3
… analog
8 Deskriptive Analyse
8.1 Visualisierung
8.1.1 Delta 1
Hier ist die Veränderung der Einsamkeit von Messung t1 zu Messung t2, aufgeteilt nach den beiden Gruppen.
ggboxplot(d_delta, x = "Geschichte", y = "loneliness_delta1",
add = "jitter")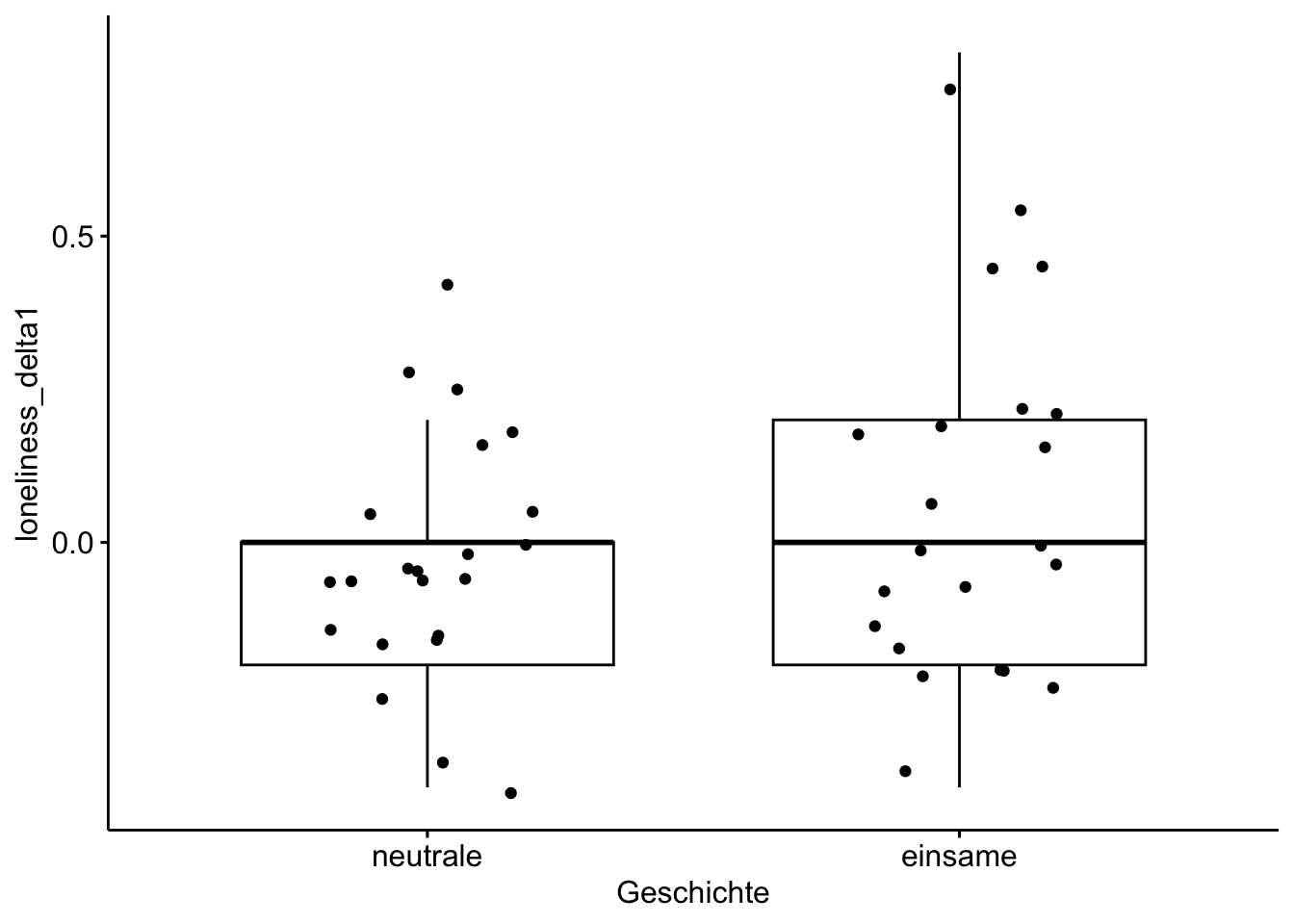
Tja, leider kein starker Effekt zu erkennen.
d_long <-
d |>
select(Proband_ID, Geschichte, starts_with("Loneliness")) |>
pivot_longer(cols = starts_with("Loneliness"),
names_to = "Messzeitpunkt",
values_to = "Einsamkeit") |>
mutate(t = as.integer(str_extract(Messzeitpunkt, "\\d"))) # Messzeitpunkt als Zahl: 1,2,3d_long |>
head() |>
kable(digits = 2)| Proband_ID | Geschichte | Messzeitpunkt | Einsamkeit | t |
|---|---|---|---|---|
| 1 | neutrale | Loneliness_mean_f1 | 1.4 | 1 |
| 1 | neutrale | Loneliness_mean_f2 | 1.2 | 2 |
| 1 | neutrale | Loneliness_mean_f3 | 1.0 | 3 |
| 2 | einsame | Loneliness_mean_f1 | 1.2 | 1 |
| 2 | einsame | Loneliness_mean_f2 | 1.8 | 2 |
| 2 | einsame | Loneliness_mean_f3 | 1.8 | 3 |
d_long |>
ggboxplot(x = "t", y = "Einsamkeit", add = "jitter",
color = "Geschichte")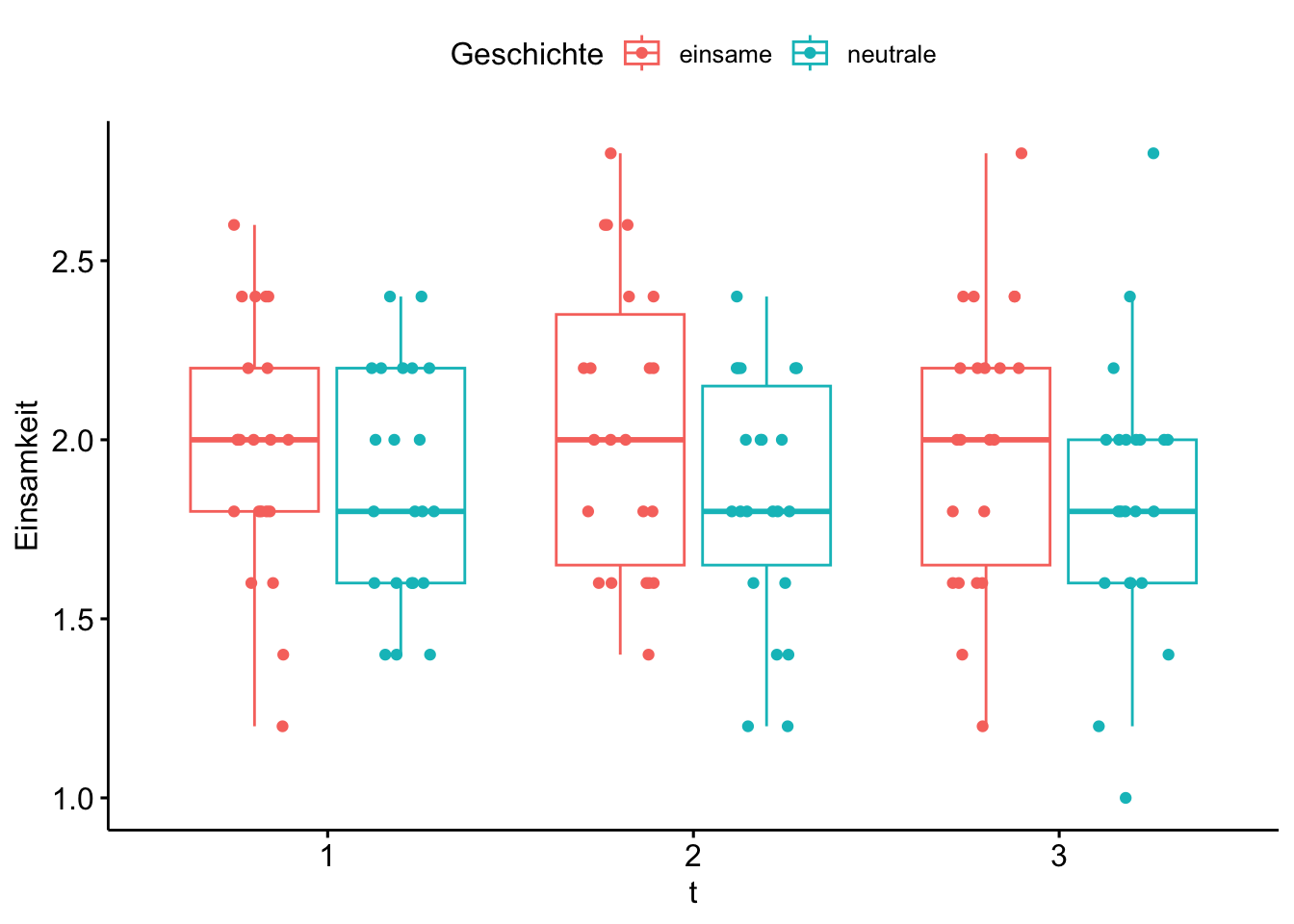
Es ist wenig Effekt zu erkennen. Interessanterweise hat die Gruppe der Einsamkeits-Intervention von vornherein einen höheren Wert in Einsamkeit. Das ist psychologisch interessant und sollte näher untersucht werden.
Andere Visualisierung:
d_long |>
ggsummarystats(x = "t",
y = "Einsamkeit",
ggfunc = ggline,
add = "median_iqr",
color = "Geschichte",
position = position_dodge(width = 0.1),
heights = c(0.6, 0.4),
caption = "Error bars show median and IQR")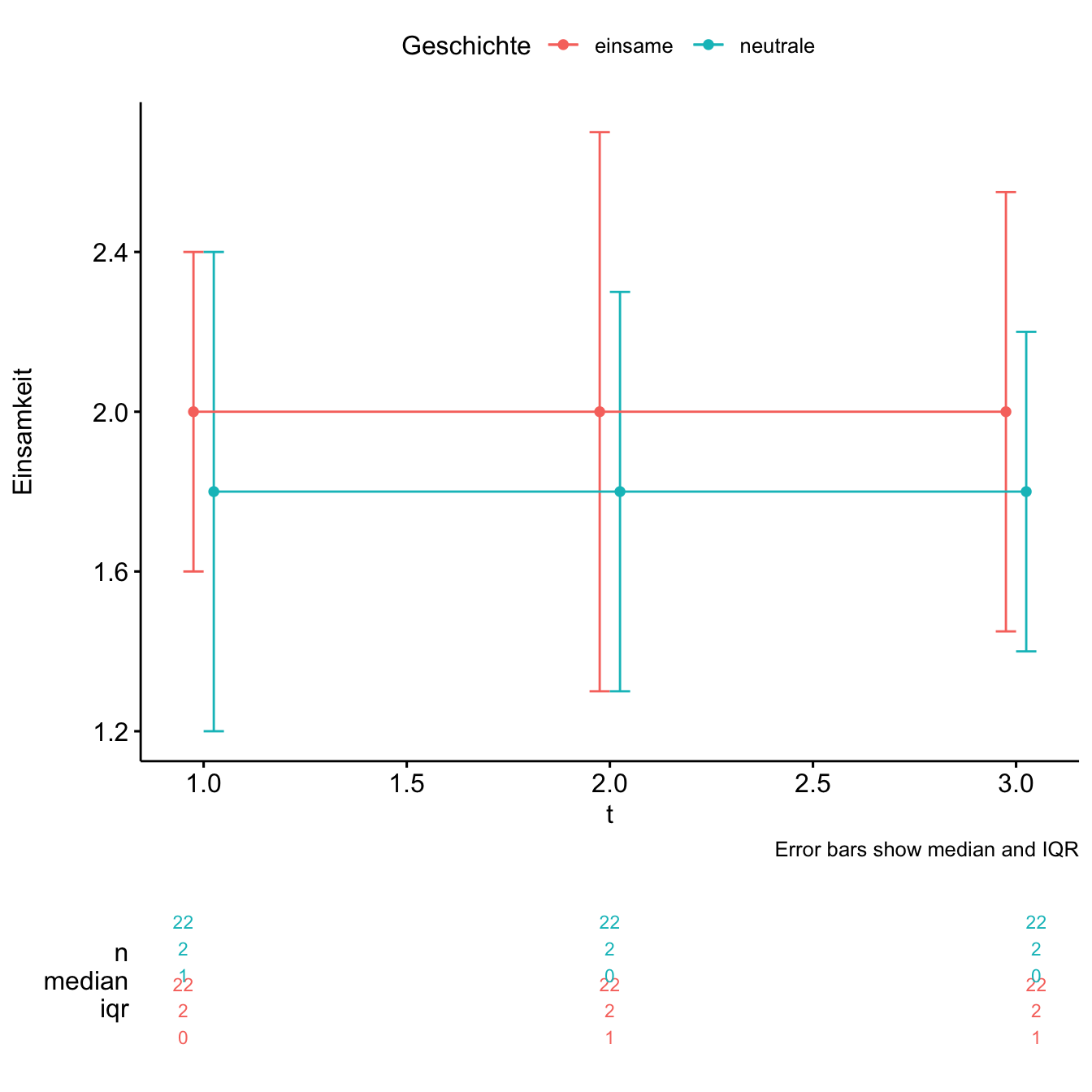
8.1.2 Delta 2 und 3
… analog
8.2 Statistiken pro Gruppe
d_delta |>
select(Geschichte, loneliness_delta1) |>
group_by(Geschichte) |>
summarise(delta1_mean = mean(loneliness_delta1),
delta1_sd = sd(loneliness_delta1))| Geschichte | delta1_mean | delta1_sd |
|---|---|---|
| einsame | 0.0727273 | 0.2930656 |
| neutrale | -0.0272727 | 0.1980424 |
9 Fazit
Veränderung-Designs analysiert man im einfachsten Fall mit Hilfe von Delta-Variablen.
In dieser Studie sind zwei UVs hintereinander gelegt. Das macht aber nichts, man kann sie getrennt voneinander analysieren.